Modellierung dynamischer Prozesse mit Differentialgleichungen (DGL) und deren Parameterschätzung mit dem Excel® Solver
Beispiel der Gelbrost (Puccinia striiformis) Dynamik am Fahnenblatt von Winterweizen
Zusammenfassung: Am Beispiel der vorhandenen Daten zur Gelbrostdynamik wird ein entsprechendes Differentialgleichungsmodell aufgestellt und die Parameterschätzung in Excel® mit Hilfe des implementierten Solver durchgeführt.
Das folgende Wirt-Parasit Modell basiert auf persönlich übermittelten Daten 1 von ca. 1997, äußerst spannende Messwerte, die in ihrer Ursprünglichkeit (wie so oft) niemals in einer Veröffentlichung Platz finden (würden). Im Rahmen dieser Parameterschätzstudie die ideale Gelegenheit, diesen Datensatz zu würdigen und dabei die Leistungsfähigkeit des Verfahrens zu demonstrieren.
In Anlehnung an die Bakterienmodelle gilt auch für grüne Pflanzenbestände annueller Pflanzen ein ähnliches dynamisches Verhalten, nach einer Anfangsphase langsamer Entwicklung folgt eine exponentielle Expansion des Bestandes, nach einem Maximum nehmen die Bestände wieder ab, um final in eine schnelle Absterbephase überzugehen. In diesem Datensatz handelt es sich nicht um einen ganzen Bestand, sondern um die Dynamik der grünen Biomasse des Fahnenblatts von Winterweizen. Während dieser Teil ohne Pilzbefall wieder durch eine analytisch lösbare Form modelliert werden kann, würde dies unter Berücksichtigung eines interagierenden Pilzbefalls nicht mehr funktionieren. (Man kann in Excel aber Modellkomponenten zusammensetzen, die eine Annäherung an die Daten ermöglichen). Wir stellen trotzdem nur das folgende numerisch zu lösende Differentialgleichungssystem zur Modellierung des Gelbrostexperiments vor:
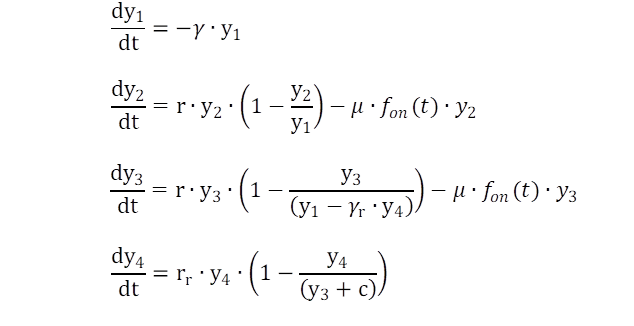
Beschreibung des Modells
Es wird, wie bei dem Bakterienansatz, mit einem zeitlich abhängigen Kapazitätsterm zur Beschreibung der abnehmenden Grünmasse begonnen (y1). DGL 2, y2 modelliert die grüne Biomasse des gesunden Fahnenblatts über die Zeit in der Form des logistischen Wachstums. Das grüne Biomasse wächst exponentiell mit der r. Limitiert wird das Wachstum wieder durch den Begrenzungsterm, der sich wiederum mit der Rate γ der DGL 1 reduziert. Wenn die biologische Zeit des Fahnenblatts abgelaufen ist, wird die Absterbesequenz mit der schon bekannten Schaltfunktion initiiert, das Blatt stirbt mit der Rate μ ab. Das Modell wiederholt das Verhalten des grünen Fahnenblatts mit ausreichender Genauigkeit (Abb. 1). Das Grundmodell wird um die Dynamik des Pilzbefalls in DGL 3, y3 erweitert: Der Kapazitätsterm des Blattes reduziert sich zusätzlich proportional zum vorhandenen Gelbrostbefall mit der Rate γr. Gleichzeitig wächst der Pilz exponentiell mit der Rate rr gemäß DGL 4, y4, limitiert wiederum durch durch die zur Verfügung stehende grüne (Rest-) Biomasse des Fahnenblatts (plus einer Konstanten c ). Die Prozesse enden mit dem Absterben des Fahnenblattes.
Obiges Modellsystem lässt sich bei gegebenen Anfangswerten sehr gut an die Daten anpassen (Abb. 1), was auf eine gewisse Plausibilität der Annahmen, die zur Modellentwicklung geführt haben, hindeutet. Die Hilfsvariable DGL 1 ist ebenfalls in Abb. 1 dargestellt (graue Linie). Die Abbildung der P. striiformis-Dynamik ist natürlich nur spekulativ (Abb.1, orange Linie), sie spiegelt die Annahmen der Modellentwicklung wider. Gleiches gilt für die gewählte Einheit auf der 2. Y-Achse. Hier könnte auch die Mycelmenge oder Uredosporenanzahl stehen. Die Anzahl der Iterationen, die Solver benötigt, lässt erkennen, wie aufwendig die Berechnungen sind. Das Anpassungsverfahren ist aber in der Lage, relativ komplexe Strukturen aufzufinden und abzubilden.
| Tab. 1: resultierende Parameterwerte für das DGL System | |||
|---|---|---|---|
| Lösung für | Abb. 1 | Abb. 2 | Abb. 3 |
| y0 für DGL 2 und 3 | 0.000716 | 0.000711 | 0.000279 |
| r | 0.365 | 0.366 | 0.402 |
| K0 für DGL 1 | 34 | 34 | 34 |
| γ | 0.004391 | 0.004667 | 0.004575 |
| tc1 | 103.3 | 96.04 | 96.3 |
| α1 | 20.06 | 36.6 | 33.43 |
| tc2 | 97.44 | 88.2 | 92.1 |
| α2 | 15.34 | 36.40 | 35.13 |
| μ | 7.06 | 10.3 | 8.29 |
| γr | 0.633 | 0.759 | 6.697 |
| y4 (t=0) | 0.159 | 0.229 | 0.645 |
| rr | 0.1589 | 0.1413 | 0.0418 |
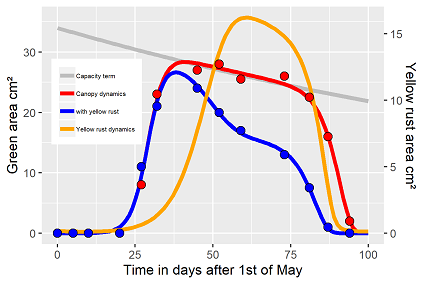
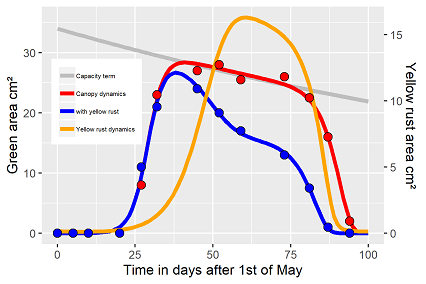
Es muss zugegeben werden, auch hier ist der Parametersatz nicht die einzige Lösung, sondern, insbesondere bei den Schaltfunktionen, von den Startwerten abhängig (Tab. 1). Man hat vom Modellprinzip mehrere Optionen: Die Schaltfunktionen beschreiben einen sanften Übergang indem man "kleine" Exponenten wählt (~15) oder einen scharfen Übergang mit "hohen" Exponenten (~35), beide führen zu einer Lösung mit ähnlicher Güte. Die Vorauswahl hat aufgrund der hohen Korrelationen innerhalb der Schaltfunktion Einfluss auf die kritischen Zeitparameter (tc.), bei dem vorgestellten Beispiel ergeben sich tc-Werte mit 103 Tagen, bzw. 98 Tagen, weit nach der Beobachtungszeit. Für Exponenten im Bereich von 35 ergeben sich tc-Werte von 96, bzw. 88 Tagen (Abb. 3). Auch wenn die Anpassungen in Abb. 3 etwas schlechter erscheinen, die tc-Werte liegen im Bereich der Beobachtungszeiten. Unabhängig von den exakten Werten, die aktive Photosynthesekapazität des infizierten Fahnenblattes ist nicht nur signifikant reduziert, sondern die Lebensdauer auch zeitlich stark verkürzt, beide Abhängigkeiten sind über die Parameter quantifizierbar. Die relativen tc-Werte beschreiben, um wie viele Tage die Photosynthese verkürzt ist, der ebenso spannende Wert γr beschreibt die Intensität des zusätzlich Verlustes an grüner Biomasse, könnte damit auch als ein Wert der Pathogenität des Pilzes, bzw. die Resistenz des zugrundeliegenden Sorte interpretiert werden. Über das Modell ist auch dieser Parameter quantitativ in weiteren Versuchen vergleichbar, falls Daten unterschiedlicher P. striiformis Stämme vorliegen würden, bzw. so ein Versuch mit verschiedenen Sorten durchgeführt würde. Ein weiteres Ergebnis: Die Dynamik des Gelbrosts ist natürlich nur das simulierte Ergebnis des Modells, es liegen diesbezüglich keine Daten vor. Unabhängig davon, es fällt auf, dass sowohl der Anfangswert y4 (t=0), als auch die Entwicklungsrate rr des Pilzes relativ hohe Schätzwerte aufweisen.
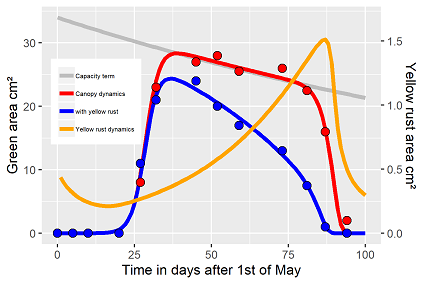
Interessanterweise führte ein alternativer Startwertvektor zu einer weiteren, aber unerwarteten und nicht ganz erklärlichen Solverlösung (Abb. 3). Zugegebenermaßen, dieses Ergebnis, insbesondere die simulierte Pilzdynamik, wurde direkt als wenig plausibel verworfen. 1. die Kurvatur des infizierten Fahnenblatts wird scheinbar ignoriert (existiert diese überhaupt?). 2.die Anpassungsprozedur führt zu doppelt so hohen Anfangswerten des Gelbrosts, die sich zuerst abbauen, da das Fahnenblatt noch gar nicht aufgetaucht ist. Im Folgenden trifft der Pilz mit entsprechender Sporenbelastung auf das sich entfaltende, frische Fahnenblattgewebe und dann entsprechend der zur Verfügung stehenden Blattfläche vermehrt, bis die Population mit dem abgestorbenen Fahnenblatt wieder zusammenbricht. Aber das Maximum liegt auf der 2. Y-Achse bei 1.5 und nicht bei 16, bzw. 14 wie in den vorherigen Beispielen. Tatsächlich ist dieses Ergebnis durchaus realistisch. Der Pilz wandert über die Vegetationsperiode hinweg sporulierend von Blattetage zur Blattetage. Es ist daher biologisch erklärbar, dass ein hohes Sporenpotential noch vor Auftauchen des Fahnenblatts vorliegt. Der resultierende Parametervektor beschreibt einen völlig andere Gelbrostdynamik als die beiden vorherigen Anpassungen. Vor allem der eventuell den Pilz charakterisierende Parameter γr ist eine 10er Potenz größer. Welche der Lösungsvarianten jetzt die "Richtige" oder "Beste" ist, ist ohne entsprechende P. striiformis Daten nicht zu lösen.
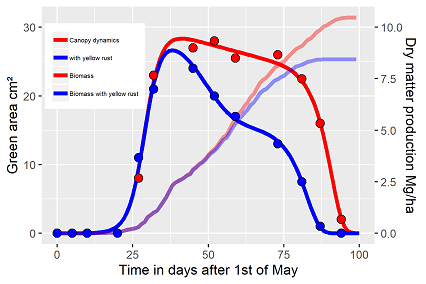
Letztendlich, welche Konsequenzen ergeben sich aus dem Gelbrostbefall? Da das Fahnenblatt ca. 95% zu der Weizenertragsbildung beiträgt, ist es die Aufgabe des Bestandsmanagements, das Fahnenblatt in seiner vollen Funktionsfähigkeit möglichst lange zu erhalten. Auf der Basis eines Beer's Law Ansatzes und entsprechenden Strahlungsdaten der zu simulierenden Zeitperiode, ergeben sich folgende relativen Trockenmassezuwächse über die Zeit (Abb. 4). Es handelt sich dabei nicht um Erträge, sondern einfach die C-Akkumulation aufgrund der photosynthetischen Aktivität. Wie Abb. 4 zeigt, die durch Lichtabsorption erzeugte Wachstumsrate ändert sich erst, nachdem das Fahnenblatt schon ca. 25% seiner Grünfläche eingebüßt hat, ca. Tag 55. Letztendlich liegen die C-Verluste im Bereich von 12% durch die pilzreduzierte Photosynthesefläche und weitere 8% folgen durch die verkürzte Lebensdauer des Fahnenblatts.
Resümee
Wie so oft bei solchen Modellen, es gibt keine eindeutige Lösung eines Systems, vor allem wenn eventuell nur Teile des Systems durch Daten verifiziert sind. Das Solver-Verfahren bietet über die simultane Parameteranpassung die Möglichkeit, die Auswahl der Möglichkeiten einzuschränken. Wichtig, wie schon in der Einleitung erwähnt, wir verwenden solche Modelle als Regressionsfunktionen für die Analyse von Feld- und Laborversuchen, d.h. um Unterschiede in den Parametervektoren den Versuchsfaktoren zuordnen zu können, sollte für alle Faktorstufen die gleichen Startwerte für den Solver verwendet werden, insbesondere wenn es sich um die Zeitschaltparameter handelt.
Literatur:
Use of in-field measurements of green leaf area and incident radiation to estimate the effects of yellow rust epidemics on the yield of winter wheat
(1997) R.J. Bryson et al., https://doi.org/10.1016/S0378-519X(97)80010-4
 Evaluation reinvented
Evaluation reinvented
