Fallbeispiel 4: Zwei- und Mehrfaktorielle Versuche
Hyperspektrale Messmethoden eignen sich auch für komplexere Versuchsanlagen. Hier kann über die kanonischen Distanzen die Intensitäten und Wechselwirkungen der einzelnen Versuchsfaktoren dargestellt und der relative Bezug interpretiert werden. Als Beispiel wird ein Sortenversuch (Topfversuch im Gewächshaus) vorgestellt, mit dem Faktor Sorte (3 Stufen: Rebensorten Müller-Thurgau (MT, anfällige Sorte), Regent (RE, resistente Sorte), Solaris (SO, resistente Sorte) 1) und dem Faktor Krankheit (Stufen: Gesund/Infiziert mit Falschem Mehltau, Plasmopara viticola), um die unterschiedliche Reaktion der Sorten auf die Pathogeninfektion aufzuzeigen. Die Pflanzen wurden zu Versuchsbeginn künstlich inokuliert. Die verwendeten Sorten unterscheiden sich hinsichtlich ihrer Resistenz/Toleranz, aber allgemein auch anhand ihrer Blattphänologie. Der Versuch war für 12 Tage angesetzt, 3 Tage nach Inokulation der obersten 3 Blätter begannen die hyperspektralen Messungen auf der Blattoberseite und wurden täglich wiederholt2.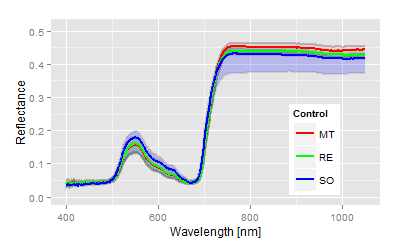
Ergebnisse
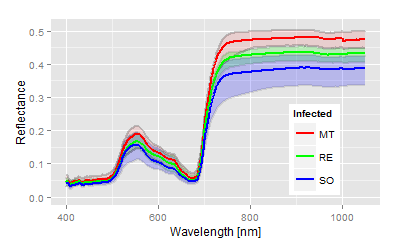
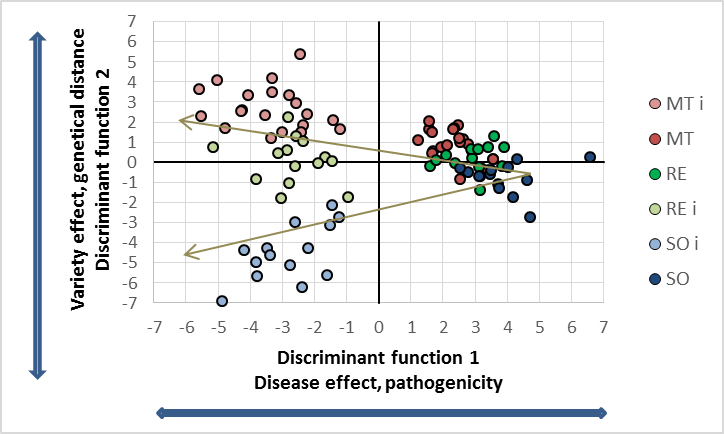
Alle statistischen Parameter sind hochsignifikant, daher werden nur die kanonischen Distanzen als Maß für die Unterschiede vorgestellt. Die verwendeten Sorten zeigen auch unbehandelt einen deutlichen Unterschied in ihren Spektren, die Blätter der Sorte Solaris sind phänologisch deutlich anders als die anderen beiden Sorten (blaues Spektrum in Abbildung links). Die Sortenunterschiede werden durch den Pathogenbefall extrem verstärkt (Abbildung der Sortenspektren rechts), so dass man anhand der Scoreverteilung auf der Diskriminanzfläche sowohl die Intensität der Infektion als auch die sortenspezifische (genetische) Response bei gegebener Pathogenität darstellen kann. Die Sorten reagieren offensichtlich und erwartungsgemäß verschieden auf den Pathogenbefall, wie es im relativen Bezug zu ersehen ist. Dabei wird keine Aussage über den Art des Unterschiedes getroffen. Tatsächlich kommt es bei MT zu akuten Gewebezerstörungen (auch ersichtlich im Spektralverlauf), während bei den Sorten Regent und Solaris das unterschiedliche Resistenzverhalten gegenüber P. viticola zum Ausdruck kommt. Der Expansionspfad definiert die Sortencharakteristika sowohl mit und ohne Falschem Mehltau, als auch die Pathogenität des verwendeten Inokulums von P. viticola.Interaktion und multiple Mittelwertvergleiche anhand der kanonischen Distanzen
Es bestehen starke Wechselwirkungen zwischen den Sorten und Pathogenbefall. Im Mittelwertvergleich sind daher gewisse Restriktionen zu beachten. Folgende Kombination der Diskriminanzanalyse sind möglich: a) Infektionskombination x alle Sorten (wie oben in der Abbildung), b) jede Sorte einzeln x Faktor Infektion (entspricht der x-Achse der Scoreverteilung), oder c) Vergleich der Sorten untereinander, einmal infiziert und nicht infiziert (entspricht der y-Achse in der Scoreverteilung). Für die Vergleiche werden die mittleren kanonischen Distanzen für alle Kombinationen dargestellt.| Mittlere kanonische Distanzen für mögliche Mittelwertvergleiche | |||||
| Kombination a, alle Faktorstufen |
Distanz | Kombination b, Pathogenität des Inokulums |
Distanz | Kombination c, genetischer Unterschied |
Distanz |
| MT x Falscher Mehltau | 6.1 | MT | 7.6 | MT vs. RE, Kontrolle | 0.83 |
| RE x Falscher Mehltau | 5.6 | RE | 12.1 | MT vs. SO, Kontrolle | 2.24 |
| SO x Falscher Mehltau | 7.7 | SO | 9.5 | RE-SO, Kontrolle | 1.42 |
| MT vs. RE, m. Pathogen | 2.72 | ||||
| MT vs. SO, m. Pathogen | 7.23 | ||||
| RE vs. SO, m. Pathogen | 4.59 | ||||
Die mittleren kanonischen Distanzen sind alle extrem hoch, nach den üblichen Kriterien ist schon ein Wert von 2-3 hochsignifikant. Diese Werte werden in diesem Versuch weit überschritten. Die dargestellte Scoreverteilung entspricht der Kombination a, die Distanzen variieren von 5 bis 8. Wenn man die Diskriminanzanalyse für jede Sorte einzeln durchführt, verstärkt sich scheinbar der biotische Stress, da die Interaktionen ausgeklammert sind. Das Ergebnis sagt nichts über Ertragsverluste etc. aus, oder das die Sorte Regent besonders stark von P. viticola betroffen wäre, sondern ergibt ein relatives Verhältnis der spezifischen Sorten in Bezug auf den biotischen Stressgrad an. Von besonderem Interesse ist die Kombination c: Die Distanzen beschreiben den genetischen Unterschied der Sorten untereinander als eine quantitative Größe, bzw. die züchterische Nähe in Bezug auf den Pathogen. Die genetischen Distanzen werden durch den Befall entsprechend des Resistenzgrades der Sorten verstärkt.
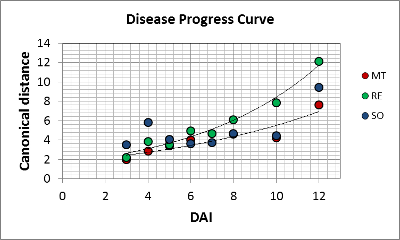
Da es sich hierbei ebenfalls um einen Zeitreihenversuch handelte, kann aus den Distanzen ein Äquivalent der Dynamik von P. viticola erstellt werden. Während sich für die Sorten MT und RE ein exponentieller Verlauf ergibt, reagiert die Sorte Solaris "scheinbar" anders. Hier starten die Messungen mit einem relativ hohen Wert von 4, d.h. es hat schon eine Reaktion auf den Pathogenbefall stattgefunden. P. viticola wird eingekapselt, es treten bis zum Tag 10 keine Veränderungen auf, erst am Tag 12 hat die hohe Dichte/Pathogenität des Inokulums die Resistenz des Wirtes gebrochen.
Die Versuche wurden zweimal mit einer deutlich geringeren Inokulumsdichte und veränderter Inkubation
wiederholt, folgen Sie dem Link zu den sehr interessanten Ergebnissen des 2. und 3. Rebenversuchs

Für ein weiteres Beispiel mit 3 Faktoren im Feldsalat folgen Sie dem zusätzlichen Link

Zusammenfassung
Auf die ANOVA Tabellen wurde hier verzichtet, da direkt mit Beginn der Messung signifikante Unterschiede vorhanden waren. Es wird vermutet, dass die Heterogenität der Sorten, bzw. Blattalter und Sorteneigenschaften schon so unterschiedlich sind, dass dieses Verfahren zwar Unterschiede aufdeckt, die aber nicht unbedingt den Effekt des Befalls beschreiben. Diese Unterschiede sind auch in den Spektren erkennbar. Die Sorte Solaris erzeugt schon in der unbehandelten Kontrolle ein anderes Spektrum (blaue Linie) als die beiden anderen Sorten.Die quantitative Analyse über die kanonischen Distanzen ermöglicht eine zutreffende Beschreibung der experimentellen Situationen und Ergebnisse. Die hohen Distanzwerte bedeuten jetzt nicht, dass es zu hohen Verlusten in den resistenten Sorten gekommen wäre. Es zeigt aber, welchen "Aufwand" die resistenten Sorten betreiben mussten, um die Infektion abzuwehren. Im Vergleich zu den weiteren Versuchen mit niedriger Pathogenität kann man ersehen, dass das überaus hohe Potential des verwendeten Inokulums die Resistenz der Sorten Regent und Solaris gebrochen hat.
Verwendet man die gegebenen Sorten mit bekannten Resistenzeigenschaften (genetische Distanz) als Standards, so würde eine neu zu testende Sorte irgendwo in dem gegebenen Bereich auf der Diskriminanzfläche auftauchen. Man kann den relativen Bezug einer neuen Sorte zu den gegebenen Standards darstellen, wobei zusätzlich der steuernde Faktor "Pathogenität" ebenfalls quantitativ dargestellt werden kann. Beide dargestellten Diskriminanzfunktionen erhalten einen biologischen Bezug, abgeleitet aus dem Experiment.
Komplexe Versuchsanlagen beinhalten den Vorteil, dass auch die Interaktionen zwischen den Faktoren berücksichtigt werden können. Die Intensität der Interaktionen zeichnen sich auch durch den Winkel der Zentroide aus. Aber die Interpretation dieser Winkel ist z.Zt. sehr schwammig, da sich die Rotation der Vektoren mit jeder Messung auf der Diskriminanzfläche ändert. Für die gerade speziell in der Pflanzenzüchtung vorkommende Situation, dass man zahlreiche neue Linien gegen einen Standard vergleicht, bietet sich in der Praxis daher an, die Kontrolle (= Vergleichssorte) als Standard auf der x-Achse zu definieren und alle Lösungen der Diskriminanzfunktionen (= Scores) relativ zur Vergleichssorte rotieren zu lassen. Das Beispiel wird auf der folgenden Seite (Phänotyisierung, Züchtung) beschrieben.
1 Sorten wurden durch das JKI Geilweiler Hof zur Verfügung gestellt.
2 In Zusammenarbeit mit Privat-Dozent Dr. E.C. Oerke,
INRES,
Institut für Pflanzenkrankheiten, Universität Bonn.
 Evaluation reinvented
Evaluation reinvented